注目の製品情報
2017/10/25
Subtractive Hybridization が Dynabeads Oligo(dT)25 で簡便に!
- タンパク・遺伝子発現解析
組織、細胞に特異的、あるいは特異的な段階において発現する遺伝子を同定する方法としてサブトラクティブハイブリダイゼーション(Subtractive Hybridization)は、非常に有用な技術です。従来の方法では「mRNAを大量に必要とする」、「偽陽性や再現不可能な結果を得る可能性が高い」、「煩雑なプロトコール」等、幾つかの問題点がありました。
Dynabeads Oligo(dT)25について
高純度のmRNAをわずか15分で単離
細胞や組織、全血などの未精製サンプルから直接高純度のmRNAをわずか15分で単離することができるDynabeads™ Oligo (dT)25は、発売以来およそ20年にわたり、mRNAの単離や精製に利用され、多くの論文に引用されています。近年ではSubtractive Hybridizationを用いた研究においても多く利用されています。
Dynabeads Oligo (dT)25 論文例
- RA Mahmoudian, et al. Applying Subtractive Hybridization Technique to Enrich and Amplify Tumor-Specific Transcripts of Esophageal Squamous Cell Carcinoma Pathology & Oncology Research. April 2017, Volume 23, Issue 2, pp 271–279
- MD Palma, et al. Suppression Subtractive Hybridization analysis provides new insights into the tomato (Solanum lycopersicum L.) response to the plant probiotic microorganism Trichoderma longibrachiatum MK1. Journal of Plant Physiology Volume 190, 15 January 2016, Pages 79-94
- Mohammad Soltany-Rezaee-Rad, et al. Overexpression of FOXO3, MYD88, and GAPDH Identified by Suppression Subtractive Hybridization in Esophageal Cancer Is Associated with Autophagy. Gastroenterology Research and Practice Volume 2014 (2014), Article ID 185035, 8 pages
- C Díez-Vives, et al. Expression of eukaryotic-like protein in the microbiome of sponges. Molecular Ecology Volume 26, Issue 5 March 2017 Pages 1432–1451
- Su MinKim, et al. Differentially expressed genes of Chenopodium amaranticolor in response to cymbidium mosaic virus infection. Virus Research Volume 223, 2 September 2016, Pages 43-51
- Zhenying Zhang, et al. Differentially‑expressed genes identified by suppression subtractive hybridization in the bone marrow hematopoietic stem cells of patients with psoriasis. Molecular Medicine Reports July 2014, Volume 10 Issue 1
Dynabeads Oligo(dT)25を用いるメリット
Dynabeads固相cDNAライブラリー(Solid-phase cDNA)
Dynabeads Oligo (dT)25では捕捉したmRNAをビーズから溶出することなく、Dynabeadsに結合したオリゴdT配列を用いて cDNA合成を行い、特定の細胞種や組織に応じた固相cDNAライブラリーを作成し、Subtractive Hybridizationに利用することができます(参考論文1-11)。
Dynabeads Oligo(dT)25を用いるメリット
- subtracted probes あるいは subtracted cDNA ライブラリーの作成が迅速、簡便にできます
- 僅かなmRNAサンプルで精製可能
- 磁石分離によりそれぞれのステップでのロスは最小限
- mRNAを高純度に濃縮
- 簡便にハイブリあるいは酵素反応に最適なバッファーに交換
Method I
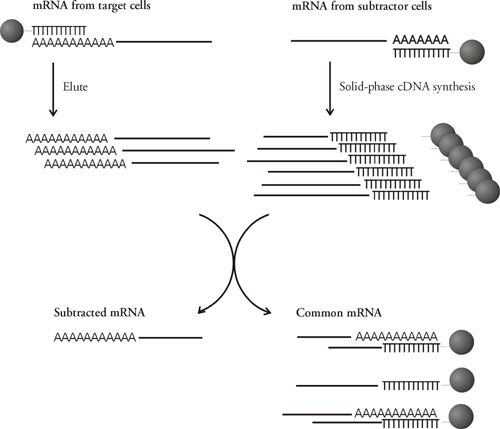
mRNA from the target material is hybridized with first-strand cDNA from the subtractor material immobilized on Dynabeads.
Dynabeadsに結合したサブトラクティブcDNAで捕捉したmRNAをマグネットで分離することによって、サブトラクトされたmRNAが上澄み液中に残ります。サブトラクションに使用したビーズは、再使用できます。
最終ハイブリダイゼーション段階が終了した後、サブトラクション固有mRNAに対して逆転写反応を行い、放射性標識cDNAを合成し、cDNAライブラリーのスクリーニング(5、10)やcDNAクローニング(9)に使用できます。 サブトラクション用のmRNA群と標的mRNA群との違いがわずかな場合には、標的mRNAを大量に得ることは難しく、Lambert(11)によるPCRを用いる方法が有用です。
Method II
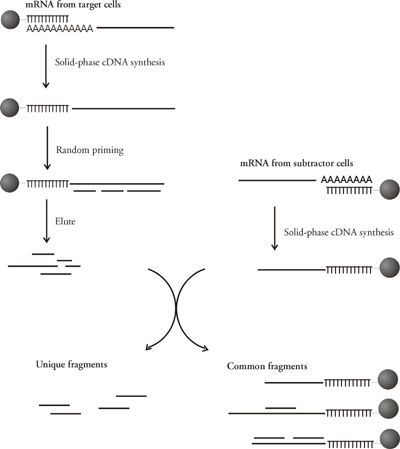
An alternative approach is to create immobilised cDNA libraries from both target and subtractor mRNA(2、4).
標的cDNAのランダムプライミングにより第2鎖cDNAを合成し、断片を溶出して、大量の固相化したサブトラクション用cDNAと混ぜ合わせます。通常の断片はアニーリングを起こして除去されますが、上清中に残った断片をプローブとして使用して、cDNAライブラリーのスクリーニングを行うことができます(2、8)。
mRNA量が限られている場合や二種類のmRNA源の類似性が高い場合には、サブトラクションのステップを複数回実施するために必要な試料がないことも考えられます。また、スクリーニングを行うためには、試料の量が不十分であることも考えられます。 このような問題は、サブトラクションしたcDNA断片を固相化したサブトラクション用cDNAに対して再アニーリングさせる方法により解決できます。
生成した二重鎖cDNAを切断し、リンカーのライゲーションを行い、PCRにより断片を増幅します(3)。
参考論文
- Camerer E et al. Binding of Factor VIIa to tissue factor on keratinocyte induces gene expression. J. Biol. Chem. 2000;275(9): 6580-6585.
- Rodriguez IR and Chader GJ. A novel method for the isolation of tissue-specific genes. Nucl.Acids.Res.1992;(13): 3528.
- CocheT, Dewez M and Beckers M-C. Generation of an unlimited supply of a subtracted probe using magnetic beads and PCR. Nucl. Acids Res. 1994;22(7): 1322-1323.
- Schoen TJ et al. Isolation of candidate genes for macular degeneration using an improved solid-phase subtractive cloning technique. Biochem. Biophys. Res. Commun.1995; 21(1)181-188.
- Aasheim H-C, Logtenberg T and Larsen F. Subtractive hybridization for the isolation of differentially expressed genes using magnetic beads. Meth. Mol. Biol. 1996; 69: 115-128.
- Leygue ER, Watson PH and Murphy LC. Identification of differentially expressed genes using minute amounts of RNA. BioTechniques 1996;21(6): 1008-1012.
- Hampson N, Hampson L and Dexter TM. Directional random oligo-nucleotide primed (DROP) global amplification of cDNA: its application to subtractive cDNA cloning. Nucl. Acids Res. 1996;24 (23): 4832-4835.
- Schraml P, Shipman R, Stulz P, Ludwig CU. cDNA subtraction library construction using a magnet-assisted subtraction technique (MAST). Trends Genet 1993; 9:70-71.
- Sharma P, Lönneborg A, Stougaard, P. PCR-based construction of subtractive cDNA library using magnetic beads. BioTechniques 1993;15(4): 610-611.
- Aasheim H-C, Deggerdal A, Smelang EB, Hornes E. A simple subtraction method for the isolation of cell-specific genes using magnetic monodisperse polymer particles. BioTechniques 1994; 16(4): 716-721.
- Lambert KN and Williamson VM. DNA library construction from small amounts of RNA using paramagnetic beads and PCR. Nucleic Acid Res 1993; 21: 775-776.